Molekulare & Experimentelle Mykobakteriologie

Um die Tuberkulose und andere Lungenerkrankungen in Zukunft erfolgreicher bekämpfen zu können, ist ein besseres Verständnis der Pathobiologie der Erreger, ihrer Evolution, Übertragung, globalen Ausbreitung und Interaktion mit dem Wirt von entscheidender Bedeutung. Hier setzt die translationale Forschungsagenda der FG MolMyc mit folgenden Schwerpunkten an: Analyse der lokalen und globalen Transmissionsdynamik, Aufklärung von Resistenz- und kompensatorischen Mechanismen, Studium der Populationsstruktur und Evolution der Mtbk Stämme und anderer Pathogene, Beschreibung der Virulenz, Physiologie und Pathobiologie von Mykobakterien, Anwendung von Hochdurchsatz Technologien in Forschung und Diagnostik, Umsetzung neuer Technologien in Ländern mit hoher Inzidenz, sowie individualisierte Therapie und Evolutionsmedizin.
Methodische Ausstattung
Die Arbeiten der FG basieren auf einer exzellenten methodischen Expertise. Next-Generation-Sequencing-(NGS-)Verfahren werden zur hochauflösenden Charakterisierung von Erregergenomen und -transkriptomen sowie zur Analyse der epigenetischen Regulation von Wirtsgenomen eingesetzt. Die methodische und technische Expertise beruht auf einer modernen Sequenzierplattform, bestehend aus Illumina NextSeq 1000 und 2000 Systemen sowie einem PacBio Vega. In den letzten Jahren wurden 10000 Proben pro Jahr sequenziert. Insgesamt sind Genomdaten von mehr als 60.000 Mtbk Stämmen und anderer Pathogene für weiterführende Analysen verfügbar. Für die bioinformatische Analyse der Sequenzdatensätze steht eine leistungsstarke IT-Infrastruktur bereit, bestehend aus einem Server mit über 700 CPU-Kernen, 3,6 TB Arbeitsspeicher und 1,2 PB Speicherplatz, die die Verarbeitung großer Datenmengen in hoher Geschwindigkeit ermöglicht. Die bioinformatischen Analyseverfahren werden kontinuierlich an die aktuellen Fragestellungen angepasst. Für die Vorhersage von Resistenzvermittelnden Mutationen, Cluster-Analysen und phylogenetischen Analysen von Mtb-Genomen stehen die in house entwickelten Plattformen MTBseq und PhyResSe.org zur Verfügung.
Im Pathobiologie-Labor stehen in vitro und in vivo Modelle zur Untersuchung klinischer Mtbk-Stämme unter relevanten Stressbedingungen (Sauerstoffdefizit/Dormanz, Antibiotika, Infektion) mit integrierten DNA-, RNA- und Proteinanalyseverfahren bereit. Ein weiterer Schwerpunkt liegt auf der Untersuchung der Langzeitevolution von Mtbk, einschließlich Antibiotikaselektion, Hysterese, Toleranz und Persister-Phänomenen in Evolutionsmedizin-Laboratorien der Sicherheitsstufen S2 und S3.
Innerhalb der Forschungsgruppe widmet sich ein Team der Implementierung moderner genombasierter Diagnostik für resistente TB in Ländern mit begrenztem Zugang zu Diagnostik und Infrastruktur. Sie bündelt Kompetenzen in der Entwicklung und Durchführung von Trainingskonzepten, der Erstellung standardisierter SOPs, sowie in Beratung und Troubleshooting und ermöglicht so eine nachhaltige, praxisnahe Umsetzung vor Ort in den Partnerländern.
Die Agenda der Forschungsgruppe wurde in vielfältige Kollaborationen und langfristige Drittmittelprojekte eingebracht. Hier sind besonders zu nennen, die Integration in das Deutsche Zentrum für Infektionsforschung (DZIF), in Netzwerkprojekte der Deutschen Forschungsgemeinschaft (RTG TransEVO, EXC 2167 PMI), des Bundesministeriums für Forschung, Technologie und Raumfahrt (BMFTR, TB-Sequel) und des BMG (Projekt im „Global Health Protection Programme“, GHPP) sowie die Beteiligung am EU-Förderprogramm „Innovative Medicines Initiative“ in den Konsortien Unite4TB und ERA4TB. Ein weiterer Meilenstein für die Forschungsgruppe Molekulare und Experimentelle Mykobakteriologie sowie die Forschungsgruppe Diagnostische Mykobakteriologie ist die erneute Ernennung des FZB zum Nationalen Referenzzentrum für Mykobakterien durch das Robert-Koch-institut (RKI) für eine Berufungsdauer bis Ende 2028. Schlüsselprojekte der gemeinsamen Arbeit im NRZ sind der Aufbau einer flächendeckenden, integrierten molekularen TB-Surveillance in Deutschland (IMS-TB, Zusammen mit dem RKI), die konstante Verbesserung der molekularen Diagnostik in Deutschland, aber auch internationale Zusammenarbeiten z.B. mit der WHO, und Studien zur Ausbreitung und Virulenz von Mykobakterien und die Unterstützung von Partnerlaboratorien beim Aufbau von diagnostischen Kapazitäten inkl. Genomsequenzierung.
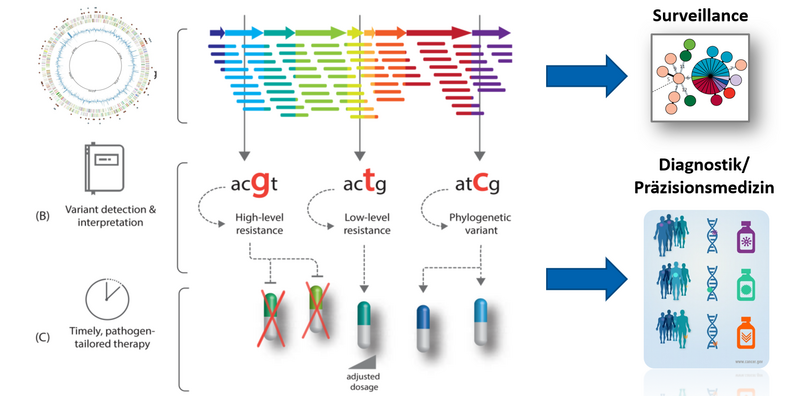
Im DZIF leitet die FG das Vorhaben „Molecular Epidemiology“. Hier werden ergänzende Studien zur aktuellen Transmissionsdynamik und Epidemiologie in verschiedenen Ländern durchgeführt. So werden longitudinale Studien für die Ausbreitung von MDR-TB Stämmen in Deutschland (Hamburg, Hannover, Frankfurt, MDR-Stämme) weitergeführt mit dem besonderen Fokus auf den Aspekt „TB und Migration“. Ein weiteres wichtiges Forschungsthema im DZIF aber auch im EXC PMI und in verschiedenen anderen internationalen Projekten ist die Weiterentwicklung von „Precision Medicine“-Konzepten für die Behandlung von Patienten mit DR-TB. Hier kommt der molekularen Diagnostik eine besondere Bedeutung zu, da für viele neuere Medikamente keine phänotypischen Verfahren für die Resistenztestung zur Verfügung stehen (siehe Abbildung).
Um Resistenzmechanismen für neue Antibiotikakandidaten aufzuklären, werden im Pathobiologie-Labor Langzeitevolutionsexperimente durchgeführt, in dem Mtbk-Stämme über einen langen Zeitraum Antibiotika ausgesetzt werden (ERA4TB). Der neuartige evolutionsbiologische Ansatz hat sich als hervorragende Methode zur Selektion von resistenten Mutanten mit einer großen phänotypischen und genetischen Vielfalt erwiesen. Die generierten Daten sollen die Etablierung von diagnostischen Verfahren vor der ersten breiten Anwendung des Wirkstoffs ermöglichen, die dann schnell zur Verfügung stehen und unmittelbar Information über etwaig entstehende Mutationen liefern können. Diese in vitro Arbeiten, werden durch große Datensätze von klinischen Mtbk-Stämmen aus Implementations- und Surveillance-Studien ergänzt. Es bestehen so ideale Voraussetzungen die Korrelation von Genotyp und Phänotyp für die Vorhersage von Resistenzen aus Genomdaten weiter zu verbessern, aber auch die Populationsstruktur, Evolution und Ausbreitung der TB-Erreger zu analysieren.
BMFTR
- 2026 – 2030 Deutsches Zentrum für Infektionsforschung (DZIF) - Translationale Einheit Tuberkulose
(TTU TB) (Co- Koordination TTU TB) Infrastruktur: Pathogenomik Labor (EpiMyc) - 2025 – 2028 DZIF-FlexFund (Co-PI) RaPaed-AIDA-TB (vormals EDCTP 2)
Schnelle und genaue Diagnose der pädiatrischen TB - eine AIDA (Assessment für innovative Diagnostik und Algorithmen für frühe und sensitive Detektion der akuten TB) Plattform Studie - 2023 – 2027 TB Sequel II (Co-PI)
Prävention, Diagnose und Behandlung von Post-Tuberkulose-Lungenerkrankung
BMG
- 2026 – 2028 Global Health Protection Program (Koordination) SeqNet+
(vormals: SeqMDRTB_NET) „Network for genomic diagnostics and surveillance on lung pathogens with a focus on drug resistant Mycobacterium tuberculosis” - 2026 - 2028 Nationales Referenzzentrum für Mycobakterien (NRZ) (Leitung)
DFG
- 2019 – 2032 PMI (Mitglied der Exekutivgruppe)
Exzellenzcluster 2167 „Präzisionsmedizin für chronische Entzündungserkrankungen“ (PMI) - 2020 – 2029 Graduiertenkolleg GRK 2501 TransEvo (Co-PI)
PhD-Programm zur translationalen evolutionären Forschung
EU
- Innovative Medicines Initiative (IMI)
- 2020 – 2026 ERA4TB (Co-PI)
European Accelerator for Tuberculosis Regime Project - 2021 – 2028 UNITE4TB (Co-PI)
Accelerating the development of new treatment regimens for tuberculosis
- 2020 – 2026 ERA4TB (Co-PI)
- Global Health EDCTP3
- 2023 – 2027 PANGenS (Koordination)
Pan-Africa Network for Genomic Surveillance of Poverty Related Disease and Emerging Pathogens - 2025 – 2031 EX-DR TB (Co-PI)
Elimination of Extensive Drug Resistance through better Regimens for TB
- 2023 – 2027 PANGenS (Koordination)
- EDCTP-Regional Networks of Excellence
- 2026 -2030 WANETAM (Co-PI)
Westafrikanisches Exzellenz-Netzwerk für TB, AIDS und Malaria
- 2026 -2030 WANETAM (Co-PI)
European Center for Disease Prevention and Control (ECDC)
- 2023 – 2026 GenEpi-BioTrain (Co-PI)
Genomic Epidemiology and Public Health Bioinformatics Trainings in the EU/EEA, the Western Balkans and Turkey
National Institutes of Health (NIH/NIAID)
- 2019 – 2027 TB Portals
Multi-national collaboration for tuberculosis data sharing and analysis to advance TB research - 2022 - 2026 R01AI170204 (Co-PI)
The effect of HIV on SARS-CoV-2 transmission and emergence of variants of concern
Leibniz-Gemeinschaft
- 2025 – 2029 Leibniz Forschungsverbund Infections (Co-PI)
IPT12: Relevance of environmental and animal reservoirs for non-tuberculous mycobacterial (NTM) disease in humans
Mikrobiologie
Standardisierte Kultursysteme zur
- Analyse von klinischen Mycobacterium tuberculosis Komplex (Mtbk)-Isolaten
- Analyse von Nicht-Tuberkulösen Mykobakterien (NTM)
- Verarbeitung größerer Probenmengen
- Anwendung verschiedener Nährmedien (fest und flüssig), inklusive Supplementen
Verfahren zur Bestimmung der minimalen Hemmkonzentration (MIC) von Mtbk-Isolaten
- REMA (Resazurin Microtiter Assay)
- EUCAST (European Committee on Antimicrobial Susceptibility Testing)
- MGIT Mycobacteria Growth Indicator Tube)
Molekularbiologie
Standardisierte Methoden für die Isolierung von mykobakterieller
- genomischer DNA
- pro-/eukaryontischer RNA
Klassische molekularbiologische Methoden für die Analyse genomischer DNA und RNA
- Agarose Gel Elektrophorese
- Southern Blot
- PCR
- Real Time PCR
- Sanger-Sequenzierungphotometrische
- fluorometrische DNA und RNA Qualitätskontrolle
- Fragmentlängenbestimmung
- Genexpressionsanalysen von Mtbk -Isolaten mittels Real-Time PCR
- Gesamtgenom Expressionsanalysen mittels: Microarray
Sequenzierungsmethoden
Next/Third Generation Sequencing Technology
- Illumina NextSeq 1000 und 2000
- PacBio Vega
- ONT Minion
- Gesamtgenomsequenzierung (auch PCR-Free)
- Expressionsanalyse mittels: RNAseq
- Sequenzierung von großen Sequenzpolymorphismen (“Regions of Difference”)
- Amplikonsequenzierung sowie überlappende Amplikon-Sequenzierung
- Methylierungssequenzierung
Bioinformatische Analyse von Sequenzdaten
- mit dem in-house entwickelten Webservice PhyResSE (phyresse.de/phyresse.org)
- mit der lokal ausführbaren in-house entwickelten Analysepipeline MTBseq (https://github.com/ngs-fzb/MTBseq_source)
- mit verschiedenen Spezialprogrammen: Seqscape, Lasergene, GenDB, Bionumerics, MBioSeq Ridom Typer
- mit Workflows für NTM- (NTMseq) und Haemophilus-Isolaten (HaemoSeq)
- Linux open source tools sowie pipelines (snakemake, nextflow)
- Analyse von Punktmutationen („Single Nucleotide Polymorphisms“)
- Analyse von großen Sequenzpolymorphismen
- Gesamtgenom Assemblierung
- Genotypisierung von MTBK-Isolaten sowie genomische Resistenzbestimmung
2026
Diricks, M, Marschall, L, Biciusca, T, Zielbauer, A-S, Kevane-Campbell, M, Kuhns, M, Andres, S, Niemann, S, Wichelhaus, TA & Wetzstein, N 2026, 'Genomic population structure, antimicrobial susceptibility, and clinical features of Mycobacterium xenopi isolates, Frankfurt, Germany, 1995-2020', JOURNAL OF CLINICAL MICROBIOLOGY, S. e0151125. https://doi.org/10.1128/jcm.01511-25
Dohál, M, Konstantynovska, O, Hromádková, M, Štrba, M, Doležalová, K, Wallenfels, J, Porvazník, I, Mäsiarová, S, Krivošová, M, Rasmussen, EM, Kunč, P, Hnilicová, J, Solovič, I, Mokrý, J, Barilar, I, Niemann, S, Diricks, M & Dvořáková, V 2026, 'Tuberculosis transmission and resistance among Ukrainian migrants and its impact on multidrug-resistant tuberculosis dynamics in high-influx host countries', BMC MICROBIOLOGY. https://doi.org/10.1186/s12866-026-04874-3
Faal, F, Top, N, Jobe, O, Colley, SM, Ayorinde, A, Mendy, A, Sarr-Kuyateh, B, Donkor, S, Antonio, M, de Jong, BC, Rachow, A, Kampmann, B, Sutherland, JS, Li, H, Blundell, TL, Campino, S, Kohl, T, Dreyer, V, Niemann, S, Pandurangan, AP, Clark, TG, Phelan, JE & Tientcheu, LD 2026, 'Genome-wide analyses of Mycobacterium tuberculosis complex isolates reveal insights into circulating lineages and drug resistance mutations in The Gambia', Scientific Reports. https://doi.org/10.1038/s41598-026-42003-2
Klingmüller, A, Zuber, S, Hoffmann, AM, Köhler, N, Suárez, I, Preßel, T, Niemann, S, Dreyer, V, Decosterd, LA, Choong, E, Schütz, B, Friesen, I & Rybniker, J 2026, 'Therapeutic drug monitoring-guided treatment of XDR TB with an RpoB I491F mutation-a case report', JAC-antimicrobial resistance, Jg. 8, Nr. 1, S. dlaf251. https://doi.org/10.1093/jacamr/dlaf251
Winkelmann, R, Blasczok, B, Winter, M, Wild, P, Vehreschild, MJGT, Kempf, VAJ, Wichelhaus, TA, Idris, R & Wetzstein, N 2026, 'Diagnostic accuracy of chip hybridization from formalin-fixed paraffin embedded bioptic specimens in a cohort with predominantly extrapulmonary mycobacterial disease', BMC MICROBIOLOGY. https://doi.org/10.1186/s12866-026-04875-2
2025
Belheouane, M, Kalsdorf, B, Niemann, S, Gaede, KI, Lange, C, Heyckendorf, J & Merker, M 2025, 'Serratia sp. traits distinguish the lung microbiome of patients with tuberculosis and non-tuberculous mycobacterial lung diseases', PLOS ONE, Jg. 20, Nr. 6, S. e0325362. https://doi.org/10.1371/journal.pone.0325362
Garcia-Prats, AJ, Garcia-Cremades, M, Cox, V, Kredo, T, Dunbar, R, Schaaf, HS, Seddon, JA, Furin, J, Achar, J, Radke, K, Sachs, T, Abubakirov, A, Ahmed, S, Akkerman, OW, Al Ani, NA, Amanullah, F, Ahmad, N, Anderson, LF, Asfaw, M, Bango, F, Bauer, T, Becerra, M, Boeree, M, Brinkmann, F, Brown, R, Brust, J, Campbell, JR, Carvalho, AC, Carvalho, I, Cegielski, JP, Centis, R, Chan, ED, Chauhan, S, Chiang, SS, Chan, P-C, D'Ambrosio, L, Dalcolmo, M, Daneilyan, N, de Vries, G, Draper, HR, endTB Study Group, Fairlie, L, Francis, JR, Franke, M, Gegia, M, Restrepo, CG, Guenther, A, Gureva, T, Haecker, B, Harausz, E, Hewison, C, Hicks, RM, Huerga, H, Hughes, J, Isaakidis, P, Syed M, K, Khan, MA, Kotrikadze, T, Kuksa, L, Lachenal, N, Lange, C, Lecca, L, Lopez-Varela, E, Lucena, S, Mariandyshev, A, Mattoo, S, Mendez-Echevarria, A, Migliori, GB, Mitnick, CD, Mohr-Holland, E, Mulanda, W, Murzabakova, T, Myrzalieve, B, Ndjeka, N, Niemann, S, Ozere, I, Padayatchi, N, Parmar, M, Parpieva, N, Manzur-Ul-Alam, M, Rybak, N, Sachdeva, KS, Salmon, K, Santiago-Garcia, B, Schaub, D, Shah, I, Shah, NS, Shah, V, Sharma, S, Shim, TS, Shin, SS, Sinha, A, Skrahina, A, Solanki, H, Solans, BP, Soriano-Arandes, A, Toktogonova, A, van der Werf , TS, Velásquez, GE, Williams, B, Yim, J-J, Savic, R & Hesseling, AC 2025, 'Characteristics of children and adolescents with multidrug-resistant and rifampicin-resistant tuberculosis and their association with treatment outcomes: a systematic review and individual participant data meta-analysis', Lancet child & adolescent health, Jg. 9, Nr. 2, S. 100-111. https://doi.org/10.1016/S2352-4642(24)00330-4
Deb, D, Heidari, ME, Kizito, B, Kesamang, M, Matila, T, Matsheka, M, Niemann, S, Shin, SS & Modongo, C 2025, 'HIV status is not associated with SARS-CoV-2 viral load and longer duration of infection among people with well-controlled HIV and high COVID-19 vaccine coverage', INTERNATIONAL JOURNAL OF INFECTIOUS DISEASES , Jg. 161, S. 108142. https://doi.org/10.1016/j.ijid.2025.108142
Diricks, M, Frank, D, Friesen, I, Niemann, S, Wichelhaus, TA, Wetzstein, N & NTMscope-Eco study group 2025, 'Global lineages of non-tuberculous mycobacteria in residential water samples from Germany', BMC MICROBIOLOGY. https://doi.org/10.1186/s12866-025-04563-7
Diricks, M, Maurer, FP, Dreyer, V, Barilar, I, Utpatel, C, Merker, M, Wetzstein, N & Niemann, S 2025, 'Genomic insights into the plasmidome of non-tuberculous mycobacteria', Genome medicine, Jg. 17, Nr. 1, S. 19. https://doi.org/10.1186/s13073-025-01443-7
Dohál, M, Wetzstein, N, Hromádková, M, Mäsiarová, S, Rasmussen, EM, Kunč, P, Škereňová, M, Porvazník, I, Solovič, I, Niemann, S, Hnilicová, J, Mokrý, J, Dvořáková, V & Diricks, M 2025, 'Diagnostics, resistance and clinical relevance of non-tuberculous mycobacteria unidentified at the species level by line probe assays: a bi-national study', Annals of clinical microbiology and antimicrobials, Jg. 24, Nr. 1, S. 14. https://doi.org/10.1186/s12941-025-00781-z
Duque-Villegas, MA, Götz, MP, Rousseau, E, Homolka, S, Niemann, S, Garred, P, Hölscher, C, Walter, K & Rosbjerg, A 2025, 'C1q and mannose-binding lectin binding and complement activation across genetically diverse Mycobacterium tuberculosis complex strains', JOURNAL OF IMMUNOLOGY. https://doi.org/10.1093/jimmun/vkaf294
Harikumar Parvathy, G*, Hertz, D*, Bhandiwad, D, Eggers, L, von Borstel, L, Behrends, J, Hein, M, Dreyer, V, Marschner, J, Orinska, Z, Niemann, S, Kaufmann, SHE, Goldmann, T, Lotter, H, Flores-Valdez, MA & Schneider, BE 2025, 'Sex Differences in Vaccine-Induced Immunity and Protection Against Mycobacterium tuberculosis', JOURNAL OF INFECTIOUS DISEASES, S. jiaf277. https://doi.org/10.1093/infdis/jiaf277 *authors contributed equally
Hillemann, D, Schweim, K, Boysen, F, Merker, M, Barilar, I, Niemann, S, Dolinger, DL, Friesen, I & Köser, CU 2025, 'Understanding systematic rifampicin resistance errors with the FluoroType MTBDR VER 2.0', INTERNATIONAL JOURNAL OF TUBERCULOSIS AND LUNG DISEASE, Jg. 29, Nr. 10, S. 469-472. https://doi.org/10.5588/ijtld.25.0150
Hoffmann, H, Utpatel, C, Iskakova, A, Ahmedov, S, Antonenka, U, Dreyer, V, Sahalchyk, E, Kadyrov, A, Corbett, C, Niemann, S & Kalmambetova, G 2025, 'Systematic investigation of baseline nosocomial transmission of tuberculosis in the Kyrgyz Republic, Central Asia', CLINICAL MICROBIOLOGY AND INFECTION . https://doi.org/10.1016/j.cmi.2025.08.001
Kessel, J, Braner, A, Diricks, M, Walther, T, Holubec, T, Werner, R, Hogardt, M, Wichelhaus, TA, Niemann, S, Moter, A, Friesen, I & Wetzstein, N 2025, 'Mycobacterium chelonae infection of a cardiovascular bioprosthesis linked to a recent outbreak', Infection. https://doi.org/10.1007/s15010-025-02534-8
Khumalo, W, Maphalala, N, Mulengwa, D, Ziyane, M, Sibandze, DB, Niemann, S, Dreyer, V, Seeger, A, Madison, M, Ness, T, Jele, T, Garcia-Basteiro, AL, Maphalala, G, DiNardo, AR, Mandalakas, AM & Kay, A 2025, 'Drug susceptibility testing for TB using the Xpert MTB/XDR assay on stool specimens', INTERNATIONAL JOURNAL OF TUBERCULOSIS AND LUNG DISEASE, Jg. 29, Nr. 4, S. 189-192. https://doi.org/10.5588/ijtld.24.0366
Költringer, F, Koreny, M, Werber, D, Heger, F, Chalupka, A, Schweiger, S, Brunner, G, Tuch, U, Karnthaler, U, Klintz, SR, Ködmön, C, Hoefer, A, Anthony, R, Kroeger, S, Domaszewska, T, Boes, L, Niemann, S, Walz, T, Kuhns, M, Jackson, S, Fitzgibbon, M, Mathys, V, Sizaire, V, Cirillo, DM, Sane Schepisi, M, Rønning, JO, Herrera-Leon, L, Cardona, P-J, Groenheit, R, Mansjö, M, Szél, V, Alm, E & Cabal, A 2025, 'Cross-border investigation of a tuberculosis outbreak in Vienna linked to a multi-country cluster among foreign-born individuals, Europe, 2021 to 2025', Eurosurveillance, Jg. 30, Nr. 33. https://doi.org/10.2807/1560-7917.ES.2025.30.33.2500565
Moyo, M, Ntshiqa, T, Hamada, Y, Copas, A, Sabi, I, Ntinginya, EN, Lalashowi, J, Matete, M, Kubeka, G, Tsope, L, Mukora, R, Mudzengi, D, Nielson, T, Lönnroth, K, Niemann, S, Rangaka, M, Velen, K, Charalambous, S, Stender, S & Minja, LT 2025, 'Community and Universal Testing for TB among close contacts of microbiologically confirmed pulmonary TB patients in two high TB burden countries: a protocol for a pragmatic cluster-randomised control trial', Trials, Jg. 26, Nr. 1, S. 285. https://doi.org/10.1186/s13063-025-08978-5
Ness, TE, Ziyane, M, Maphalala, N, Seeger, A, Vasiliu, A, Khumalo, W, Thunzini, M, Dlamini, S, Maphalala, G, Gascua, C, Lange, C, Meyer, S, Inman, B, Dreyer, V, Utpatel, C, Niemann, T, DiNardo, A, Kay, A, Niemann, S & Mandalakas, A 2025, 'Screening and targeted sequencing of stool for microbiologic confirmation and drug resistance determination in paucibacillary tuberculosis', PLOS global public health, Jg. 5, Nr. 11, S. e0005243. https://doi.org/10.1371/journal.pgph.0005243
Ntshiqa, T, Fasanmi, A, Nagudi, J, Tsope, L, Copas, A, Stender, S, Sabi, I, Ntinginya, EN, Lalashowi, J, Matete, M, Budiaki, L, Mahlalela, M, Kisinda, A, Mudzengi, D, Minja, LT, Chirwa, T, Lönnroth, K, Niemann, S, Dreyer, V, Charalambous, S, Velen, K, Hamada, Y & Rangaka, M 2025, 'Evaluation of QIAreach QuantiFERON-TB lateral-flow nanoparticle fluorescence assay for TB infection diagnosis among TB household contacts in three high-burden settings', PLOS ONE, Jg. 20, Nr. 9, S. e0332125. https://doi.org/10.1371/journal.pone.0332125
Ntshiqa, T, Nagudi, J, Hamada, Y, Copas, A, Stender, S, Sabi, I, Ntinginya, EN, Lalashowi, J, Matete, M, Ntshamane, K, Morojele, I, Ngobeni, M, Mudzengi, D, Minja, LT, Chirwa, T, Lönnroth, K, Dreyer, V, Niemann, S, Rangaka, M, Charalambous, S & Velen, K 2025, 'Risk factors associated with TB infection among household contacts of microbiologically confirmed pulmonary TB patients in three high TB burden countries', THE JOURNAL OF INFECTIOUS DISEASES. https://doi.org/10.1093/infdis/jiaf320
Nurjadi, D, Schulenburg, H, Niemann, S, Zinkernagel, AS & Rupp, J 2025, 'Are we overlooking the obvious? Bacterial evolution is at the heart of antimicrobial resistance', The Lancet. Microbe, S. 101069. https://doi.org/10.1016/j.lanmic.2024.101069
Palme, PR, Grover, S, Abdelaziz, R, Mann, L, Kany, AM, Ouologuem, L, Bartel, K, Sonnenkalb, L, Reiling, N, Hirsch, AKH, Schnappinger, D, Rubinstein, JL, Imming, P & Richter, A 2025, 'Design, Synthesis, and Biological Evaluation of Mono- and Diamino-Substituted Squaramide Derivatives as Potent Inhibitors of Mycobacterial Adenosine Triphosphate (ATP) Synthase', JOURNAL OF MEDICINAL CHEMISTRY. https://doi.org/10.1021/acs.jmedchem.5c02284
Petersen, S, Diricks, M, Utpatel, C, Schulenburg, H & Merker, M 2025, 'Evolution of beta-lactam resistance causes fitness reductions and several cases of collateral sensitivities in the human pathogen Haemophilus influenzae', ANTIMICROBIAL AGENTS AND CHEMOTHERAPY, S. e0057625. https://doi.org/10.1128/aac.00576-25
Pohl, J, Gröschel, MI, Neumann, N, Tillmann, A, Waldeck, S, Schneider, T, Overhoff, D, Lange, SG, Bucksch, T, Peuker, R, Rauschning, D, Scheumann, G, Scheit, L, Schreiner, M, Wenzel, W, Kehe, K, Friesen, I, Dreyer, V, Niemann, S, Sander, L-E, Witzenrath, M, Nouailles, G, Zobel, CM & Retro-TB-Bw-Study Group 2025, 'Active case finding for tuberculosis among Ukrainian soldiers at the German Armed Forces', The European respiratory journal. https://doi.org/10.1183/13993003.00728-2025
Protic, D, Bascarevic, D, Dimitrijevic, S, Pesovic, J, Nikolic, V, Nikolic, S, Novicevic, V, Markovic, J, Arandjelovic, I, Savic-Pavicevic, D, Diricks, M, Belheouane, M & Merker, M 2025, 'Microbiome modulation and behavioural improvements in children with fragile X syndrome following probiotic intake: A pilot study', Scientific Reports. https://doi.org/10.1038/s41598-025-29896-1
Quansah, N, Arriaga, MB, Calderon, RI, Shin, S, Molldrem, S, Niemann, S & Ugarte-Gil, C 2025, 'Flying blind: urgency for drug-resistant testing for new tuberculosis drugs', The Lancet. Microbe, S. 101106. https://doi.org/10.1016/j.lanmic.2025.101106
Skouvig Pedersen, O, Butova, T, Miasoiedov, V, Feshchenko, Y, Kuzhko, M, Niemann, S, Rosenthal, A, Grinev, A, Rosenfeld, G, Hoppes, MD, Kilmnick, J, Crudu, V, Ciobanu, N, Codreanu, A, Toxanbayeva, B, Chingissova, L, Yurko, K, Kucheriavchenko, V, Vekshyn, V, Vashakidze, S, Shubladze, N, Avaliani, Z, Kadyrov, A, Kalmambetova, G, Sydykova, M, Ghita, E, Grecu, VI, Miulescu, AM, Wejse, CM, Fløe, A, Dahl, VN & Butov, D 2025, 'Sex differences in risk factors for unsuccessful tuberculosis treatment outcomes in Eastern Europe from 2020 to 2022: a multi-country retrospective cohort study', The Lancet regional health. Europe, Jg. 55, S. 101354. https://doi.org/10.1016/j.lanepe.2025.101354
Sonnenkalb, L, Trubenová, B, Regoes, RR, Merker, M & Niemann, S 2025, 'The mutant selection window of moxifloxacin and bedaquiline resistant Mycobacterium tuberculosis', Journal of infection, S. 106523. https://doi.org/10.1016/j.jinf.2025.106523
Szél, V, Phelan, JE, Georghiou, SB, Dolinger, DL, Clark, TG, Niemann, S, Lőrinczi, LK & Köser, CU 2025, 'The ahpC c-54t compensatory mutation is not always a valid surrogate for isoniazid resistance in Mycobacterium tuberculosis', ANTIMICROBIAL AGENTS AND CHEMOTHERAPY, S. e0026525. https://doi.org/10.1128/aac.00265-25
Trauth, J, Laspoulas, A, Dakischew, O, Gentil, K, Dreyer, V, Kuhns, M, Friesen, I & Herold, S 2025, 'Bedaquiline-Pretomanid-Linezolid as a Bridging Therapy for Adverse Events in Drug-Sensitive Tuberculosis', American journal of respiratory and critical care medicine. https://doi.org/10.1164/rccm.202501-0150RL
Tukvadze, N, Mashatole, S, Jacobs, A, Ngwanto, T, Heinrich, N, Van Rie, A, Crudu, V, Tudor, E, Goginashvili, L, Ibraim, E, Khosa, C, Howell, P, Rajaram, S, Naidoo, K, Niemann, S, Reither, K & Wallis, RS 2025, 'Drtb-hdt: a randomized controlled trial of two adjunctive host-directed therapies in rifampin-resistant tuberculosis', BMC INFECTIOUS DISEASES , Jg. 25, Nr. 1, S. 768. https://doi.org/10.1186/s12879-025-11072-5
Wang, Q, Barilar, I, Minin, VM, Modongo, C, Moonan, PK, Finlay, A, Boyd, R, Oeltmann, JE, Molefi, TL, Zetola, NM, Brewer, TF, Niemann, S & Shin, SS 2025, 'Phylodynamic analysis reveals disparate transmission dynamics of Mycobacterium tuberculosis complex lineages in Botswana', Scientific Reports. https://doi.org/10.1038/s41598-025-31425-z
Wei, KC, Purushothaman, S, Azzato, F, Baker, KS, Birkeland, KW, Brunet, S, Campos, J, Connor, TR, Giske, CG, Greub, G, Hallbäck, ET, Harmsen, D, Hodcroft, EB, Holden, MTG, Jacob, J, Kahles, A, Lagos, AC, Lipworth, S, Loo, E, Miotto, P, Neher, RA, Neves, A, Ready, D, Roloff, T, Rooney, A, Rousseau, E, Schrenzel, J, Sundqvist, M, Viegas, S, Wegner, F, Aamot, HV, Niemann, S, Williamson, DA, Mölling, P & Egli, A 2025, 'Conference report: The second Bacterial Genome Sequencing Pan-European Network conference', Microbes and Infection, S. 105557. https://doi.org/10.1016/j.micinf.2025.105557
2024
Adegbite, BR, Dreyer, V, Agbo, JBPAA, Mevyann, RC, Mfoumbi, GARI, Ndanga, MED, Biyogho, CM, Edoa, JR, M'Baidiguim, FB, Ndong, AROO, Alabi, AS, Kremsner, PG, Adegnika, AA, Niemann, S & Grobusch, MP 2025, 'Clinical epidemiology, genetic diversity, and drug susceptibility patterns by whole genome sequencing of Mycobacterium tuberculosis complex isolates in Gabon from 2012 to 2022', IJID regions, Jg. 14, S. 100501. https://doi.org/10.1016/j.ijregi.2024.100501
Barilar, I, Fernando, T, Utpatel, C, Abujate, C, Madeira, CM, José, B, Mutaquiha, C, Kranzer, K, Niemann, T & Ismael, N et al. 2024, 'Emergence of bedaquiline-resistant tuberculosis and of multidrug-resistant and extensively drug-resistant Mycobacterium tuberculosis strains with rpoB Ile491Phe mutation not detected by Xpert MTB/RIF in Mozambique: a retrospective observational study', Lancet Infectious Diseases, Jg. 24, Nr. 3, S. 297-307. https://doi.org/10.1016/S1473-3099(23)00498-X
Blankson, HNA, Kamara, RF, Barilar, I, Andres, S, Conteh, OS, Dallenga, T, Foray, L, Maurer, F, Kranzer, K & Utpatel, C et al. 2024, 'Molecular determinants of multidrug-resistant tuberculosis in Sierra Leone', Microbiology spectrum, Jg. 12, Nr. 3, S. e0240523. https://doi.org/10.1128/spectrum.02405-23
CRyPTIC Consortium 2024, 'Quantitative drug susceptibility testing for Mycobacterium tuberculosis using unassembled sequencing data and machine learning', PLoS Computational Biology , Jg. 20, Nr. 8, S. e1012260. https://doi.org/10.1371/journal.pcbi.1012260
CRyPTIC Consortium 2024, 'Quantitative measurement of antibiotic resistance in Mycobacterium tuberculosis reveals genetic determinants of resistance and susceptibility in a target gene approach', Nature communications, Jg. 15, Nr. 1, S. 488. https://doi.org/10.1038/s41467-023-44325-5
Danchuk, SN, Solomon, OE, Kohl, TA, Dreyer, V, Barilar, I, Utpatel, C, Niemann, S, Soolingen, DV, Anthony, R & van Ingen, J et al. 2024, 'Challenging the gold standard: the limitations of molecular assays for detection of Mycobacterium tuberculosis heteroresistance', Thorax, Jg. 79, Nr. 7, S. 670-675. https://doi.org/10.1136/thorax-2023-220202
Diricks, M, Petersen, S, Bartels, L, Lâm, T-T, Claus, H, Bajanca-Lavado, MP, Hauswaldt, S, Stolze, R, Vázquez, OJ, Utpatel, C, Niemann, S, Rupp, J, Wohlers, I & Merker, M 2024, 'Revisiting mutational resistance to ampicillin and cefotaxime in Haemophilus influenzae', Genome medicine, Jg. 16, Nr. 1, S. 140. https://doi.org/10.1186/s13073-024-01406-4
Dreyer, V, Sonnenkalb, L, Diricks, M, Utpatel, C, Barilar, I, Mohr, V, Niemann, S, Kohl, TA & Merker, M 2024, 'Use of Whole Genome Sequencing for Mycobacterium tuberculosis Complex Antimicrobial Susceptibility Testing: From Sequence Data to Resistance Profiles', Methods in Molecular Biology, Jg. 2833, S. 195-210. https://doi.org/10.1007/978-1-0716-3981-8_18
Dyer, NP, Päuker, B, Baxter, L, Gupta, A, Bunk, B, Overmann, J, Diricks, M, Dreyer, V, Niemann, S, Holt, KE, Rahman, M, Brown, PE, Stark, R, Zhou, Z, Ott, S & Nübel, U 2024, 'EnteroBase in 2025: exploring the genomic epidemiology of bacterial pathogens', NUCLEIC ACIDS RESEARCH. https://doi.org/10.1093/nar/gkae902
Friesen, I, Dreyer, V, Klingmüller, A, Zuber, S, Hoffmann, AM, Suárez, I, Schütz, B, Preßel, T, Andres, S & Niemann, S et al. 2024, 'First detection of a Mycobacterium tuberculosis XDR clinical isolate harbouring an RpoB I491F mutation in a Ukrainian patient treated in Germany, October 2023', Eurosurveillance, Jg. 29, Nr. 28. https://doi.org/10.2807/1560-7917.ES.2024.29.28.2400420
Friesen, I, Saluzzo, F, Groenheit, R, Aubry, A, Anthony, R, Niemann, S, Mathys, V, Cirillo, DM & TB European Reference Laboratory Network (ERLTB-Net-3) 2024, 'Re: 'Availability of drugs and resistance testing for BPaLM regimen for rifampicin-resistant tuberculosis in Europe' by Lange et al', CLINICAL MICROBIOLOGY AND INFECTION , Jg. 30, Nr. 9, S. 1204-1206. https://doi.org/10.1016/j.cmi.2024.06.001
Germuskova, Z, Sosa, E, Lagos, AC, Aamot, HV, Beale, MA, Bertelli, C, Björkmann, J, Couto, N, Feige, L & Greub, G et al. 2024, 'Conference report: The first Bacterial Genome Sequencing Pan-European Network Conference', Microbes and Infection, S. 105410. https://doi.org/10.1016/j.micinf.2024.105410
Götz, MP, Duque Villegas, MA, Fageräng, B, Kerfin, A, Skjoedt, M-O, Garred, P & Rosbjerg, A 2024, 'Transient Binding Dynamics of Complement System Pattern Recognition Molecules on Pathogens', JOURNAL OF IMMUNOLOGY, Jg. 212, Nr. 9, S. 1493-1503. https://doi.org/10.4049/jimmunol.2300768
Gröschel, MI, Pérez-Llanos, FJ, Diel, R, Vargas, R, Escuyer, V, Musser, K, Trieu, L, Meissner, JS, Knorr, J & Klinkenberg, D et al. 2024, 'Differential rates of Mycobacterium tuberculosis transmission associate with host-pathogen sympatry', Nature Microbiology, Jg. 9, Nr. 8, S. 2113-2127. https://doi.org/10.1038/s41564-024-01758-y
Günther, G, Mhuulu, L, Diergaardt, A, Dreyer, V, Moses, M, Anyolo, K, Ruswa, N, Claassens, M, Niemann, S & Nepolo, E 2024, 'Bedaquiline Resistance after Effective Treatment of Multidrug-Resistant Tuberculosis, Namibia', EMERGING INFECTIOUS DISEASES, Jg. 30, Nr. 3, S. 568-571. https://doi.org/10.3201/eid3003.240134
Hansen, J, Kolbe, K, König, IR, Scherließ, R, Hellfritzsch, M, Malm, S, Müller-Loennies, S, Zallet, J, Hillemann, D, Wiesmüller, K-H, Herzmann, C, Brandenburg, J & Reiling, N 2022, 'Lipobiotin-capture magnetic bead assay for isolation, enrichment and detection of Mycobacterium tuberculosis from saliva', PLOS ONE, Jg. 17, Nr. 7, S. e0265554. https://doi.org/10.1371/journal.pone.0265554
Idris, R, Zielbauer, A-S, Koepsell, J, Kloka, J & Wetzstein, N 2024, 'Extracorporeal membrane oxygenation (ECMO) in patients with tuberculosis: systematic review and meta-analysis of 43 cases', BMC PULMONARY MEDICINE, Jg. 24, Nr. 1, S. 47. https://doi.org/10.1186/s12890-023-02715-x
Köser, CU, Miotto, P, Ismail, N, Anthony, RM, Utpatel, C, Merker, M, Niemann, S, Tahseen, S, Rigouts, L & Rodrigues, C et al. 2024, 'A composite reference standard is needed for bedaquiline antimicrobial susceptibility testing for Mycobacterium tuberculosis complex', The European respiratory journal, Jg. 64, Nr. 1. https://doi.org/10.1183/13993003.00391-2024
Larsson, L, Corbett, C, Kalmambetova, G, Utpatel, C, Ahmedov, S, Antonenka, U, Iskakova, A, Kadyrov, A, Kohl, TA & Barilar, V et al. 2024, 'Whole-genome sequencing drug susceptibility testing is associated with positive MDR-TB treatment response', INTERNATIONAL JOURNAL OF TUBERCULOSIS AND LUNG DISEASE, Jg. 28, Nr. 10, S. 494-499. https://doi.org/10.5588/ijtld.24.0052
Machado, E, Vasconcellos, S, Gomes, L, Catanho, M, Ramos, J, de Carvalho, L, Goldenberg, T, Redner, P, Caldas, P & Campos, C et al. 2024, 'Phylogenomic and genomic analysis reveals unique and shared genetic signatures of Mycobacterium kansasii complex species', Microbial genomics, Jg. 10, Nr. 7. https://doi.org/10.1099/mgen.0.001266
Meiwes, L, Kontsevaya, I, Chesov, D, Kulciţkaia, S, Dreyer, V, Hillemann, D, Dlamini, Q, Williams, C, Barer, M & Brinkmann, F et al. 2024, 'Whispers in the wind: Face mask sampling for Mycobacterium tuberculosis detection in children with pulmonary tuberculosis', JOURNAL OF INFECTIOUS DISEASES. https://doi.org/10.1093/infdis/jiae282
Mohr, V, Sonnenkalb, L, Utpatel, C, Barilar, I, Diricks, M, Dreyer, V, Niemann, S, Kohl, TA & Merker, M 2024, 'Use of Whole Genome Sequencing for Mycobacterium tuberculosis Complex Antimicrobial Susceptibility Testing', Methods in Molecular Biology, Jg. 2833, S. 185-193. https://doi.org/10.1007/978-1-0716-3981-8_17
Olbrich, L, Franckling-Smith, Z, Larsson, L, Sabi, I, Ntinginya, NE, Khosa, C, Banze, D, Nliwasa, M, Corbett, EL, Semphere, R, Verghese, VP, Michael, JS, Ninan, MM, Saathoff, E, McHugh, TD, Razid, A, Graham, SM, Song, R, Nabeta, P, Trollip, A, Nicol, MP, Hoelscher, M, Geldmacher, C, Heinrich, N, Zar, HJ & RaPaed-AIDA-TB consortium 2024, 'Sequential and parallel testing for microbiological confirmation of tuberculosis disease in children in five low-income and middle-income countries: a secondary analysis of the RaPaed-TB study', Lancet Infectious Diseases. https://doi.org/10.1016/S1473-3099(24)00494-8
Phelan, JE, Utpatel, C, Ismail, N, Cortes, T, Niemann, S, Cirillo, DM, Schön, T, Miotto, P & Köser, CU 2024, 'Careful classification of potential bedaquiline resistance mutations is critical when analysing their clinical impact', INTERNATIONAL JOURNAL OF TUBERCULOSIS AND LUNG DISEASE, Jg. 28, Nr. 6, S. 312-313. https://doi.org/10.5588/ijtld.24.0083
Rachwal, N, Idris, R, Dreyer, V, Richter, E, Wichelhaus, TA, Niemann, S, Wetzstein, N & Götsch, U 2024, 'Pathogen and host determinants of extrapulmonary tuberculosis among 1035 patients in Frankfurt am Main, Germany, 2008-2023', CLINICAL MICROBIOLOGY AND INFECTION . https://doi.org/10.1016/j.cmi.2024.11.009
Rupasinghe, P, Reenaers, R, Vereecken, J, Mulders, W, Cogneau, S, Merker, M, Niemann, S, Vally Omar, S, Rigouts, L & Köser, CU et al. 2024, 'Refined understanding of the impact of the Mycobacterium tuberculosis complex diversity on the intrinsic susceptibility to pretomanid', Microbiology spectrum, Jg. 12, Nr. 3, S. e0007024. https://doi.org/10.1128/spectrum.00070-24
Shavuka, O, Iipumbu, E, Boois, L, Günther, G, Hoddinott, G, Lin, H-H, Nepolo, E, Niemann, S, Ruswa, N & Seddon, J et al. 2024, 'Enhanced active case finding of drug-resistant tuberculosis in Namibia: a protocol for the hotspots, hospitals, and households (H3TB) study', BMJ open, Jg. 14, Nr. 2, S. e082665. https://doi.org/10.1136/bmjopen-2023-082665
Svensson, E, Ketelsen, H, Andres, S, Folkvardsen, DB, Hillemann, D, Conteh, O, Norman, A, Niemann, S, Lillebaek, T & Kuhns, M 2024, 'Dual-centre evaluation of the FluoroType MTBDR version 2 assay for detection of Mycobacterium tuberculosis complex and resistance-conferring mutations in pulmonary and extrapulmonary samples from Denmark, Germany and Sierra Leone', CLINICAL MICROBIOLOGY AND INFECTION , Jg. 30, Nr. 8, S. 1055-1060. https://doi.org/10.1016/j.cmi.2024.04.006
Utpatel, C, Zavaleta, M, Rojas-Bolivar, D, Mühlbach, A, Picoy, J, Portugal, W, Esteve-Solé, A, Alsina, L, Miotto, P & Bartholomeu, DC et al. 2024, 'Prison as a driver of recent transmissions of multidrug-resistant tuberculosis in Callao, Peru: a cross-sectional study', Lancet regional health. Americas, Jg. 31, S. 100674. https://doi.org/10.1016/j.lana.2024.100674
Wetzstein, N, Diricks, M, Andres, S, Kuhns, M, Marschall, L, Biciusca, T, Smaczny, C, Friesen, I, Niemann, S & Wichelhaus, TA 2024, 'Genomic diversity and clinical relevance of Mycobacterium simiae', ERJ Open Research, Jg. 10, Nr. 2, 00773-2023. https://doi.org/10.1183/23120541.00773-2023
Wetzstein, N, Diricks, M, Anton, TB, Andres, S, Kuhns, M, Kohl, TA, Schwarz, C, Lewin, A, Kehrmann, J & Kahl, BC et al. 2024, 'Clinical and genomic features of Mycobacterium avium complex: a multi-national European study', Genome medicine, Jg. 16, Nr. 1, S. 86. https://doi.org/10.1186/s13073-024-01359-8
2023
Baker, CR, Barilar, I, de Araujo, LS, Rimoin, AW, Parker, DM, Boyd, R, Tobias, JL, Moonan, PK, Click, ES, Finlay, A, Oeltmann, JE, Minin, VN, Modongo, C, Zetola, NM, Niemann, S & Shin, SS 2023, 'Use of High-Resolution Geospatial and Genomic Data to Characterize Recent Tuberculosis Transmission, Botswana', EMERGING INFECTIOUS DISEASES, Jg. 29, Nr. 5, S. 977-987. https://doi.org/10.3201/eid2905.220796
Botelho, J, Tüffers, L, Fuss, J, Buchholz, F, Utpatel, C, Klockgether, J, Niemann, S, Tümmler, B & Schulenburg, H 2023, 'Phylogroup-specific variation shapes the clustering of antimicrobial resistance genes and defence systems across regions of genome plasticity in Pseudomonas aeruginosa', EBioMedicine, Jg. 90, S. 104532. https://doi.org/10.1016/j.ebiom.2023.104532
Chesov, E, Chesov, D, Reimann, M, Dreyer, V, Utpatel, C, Gröschel, MI, Ciobanu, N, Crudu, V, Lange, C, Heyckendorf, J & Merker, M 2023, 'Impact of Mycobacterium tuberculosis strain type on multidrug-resistant tuberculosis severity, Republic of Moldova', Journal of infection, Jg. 87, Nr. 6, S. 588-591. https://doi.org/10.1016/j.jinf.2023.10.001
Comprehensive Resistance Prediction for Tuberculosis: an International Consortium, Sonnenkalb, L, Carter, JJ, Spitaleri, A, Iqbal, Z, Hunt, M, Malone, KM, Utpatel, C, Cirillo, DM, Rodrigues, C, Nilgiriwala, KS, Fowler, PW, Merker, M & Niemann, S 2023, 'Bedaquiline and clofazimine resistance in Mycobacterium tuberculosis: an in-vitro and in-silico data analysis', The Lancet. Microbe, Jg. 4, Nr. 5, S. e358-e368. https://doi.org/10.1016/S2666-5247(23)00002-2
de Araujo, L, Cabibbe, AM, Mhuulu, L, Ruswa, N, Dreyer, V, Diergaardt, A, Günther, G, Claassens, M, Gerlach, C, Utpatel, C, Cirillo, DM, Nepolo, E & Niemann, S 2023, 'Implementation of targeted next-generation sequencing for the diagnosis of drug-resistant tuberculosis in low-resource settings: a programmatic model, challenges, and initial outcomes', Frontiers in public health, Jg. 11, S. 1204064. https://doi.org/10.3389/fpubh.2023.1204064
Diel, R, Meywald-Walther, K, Schwarzbach, C, Voss, K, Dreyer, V & Niemann, S 2023, 'Risk of tuberculosis transmission by children in Hamburg, Germany', RESPIRATORY MEDICINE , Jg. 209, S. 107152. https://doi.org/10.1016/j.rmed.2023.107152
Economou Lundeberg, E, Andersson, V, Wijkander, M, Groenheit, R, Mansjö, M, Werngren, J, Cortes, T, Barilar, I, Niemann, S, Merker, M, Köser, CU & Davies Forsman, L 2023, 'In vitro activity of new combinations of β-lactam and β-lactamase inhibitors against the Mycobacterium tuberculosis complex', Microbiology spectrum, Jg. 11, Nr. 5, S. e0178123. https://doi.org/10.1128/spectrum.01781-23
ERA4TB consortium, van Wijk, RC, Lucía, A, Sudhakar, PK, Sonnenkalb, L, Gaudin, C, Hoffmann, E, Dremierre, B, Aguilar-Ayala, DA, Molin, MD, Rybniker, J, de Giorgi, S, Cioetto-Mazzabò, L, Segafreddo, G, Manganelli, R, Degiacomi, G, Recchia, D, Pasca, MR, Simonsson, USH & Ramón-García, S 2023, 'Implementing best practices on data generation and reporting of Mycobacterium tuberculosis in vitro assays within the ERA4TB consortium', iScience, Jg. 26, Nr. 4, S. 106411. https://doi.org/10.1016/j.isci.2023.106411
Gries, R, Dal Molin, M, Chhen, J, van Gumpel, E, Dreyer, V, Niemann, S & Rybniker, J 2023, 'Characterization of Two Novel Inhibitors of the Mycobacterium tuberculosis Cytochrome bc1 Complex', ANTIMICROBIAL AGENTS AND CHEMOTHERAPY, Jg. 67, Nr. 7, S. e0025123. https://doi.org/10.1128/aac.00251-23
Ismael, N, van Wyk, S, Tegally, H, Giandhari, J, San, JE, Moir, M, Pillay, S, Utpatel, C, Singh, L, Naidoo, Y, Ramphal, U, Mabunda, N, Abílio, N, Arnaldo, P, Xavier, J, Amoako, DG, Everatt, J, Ramphal, Y, Maharaj, A, de Araujo, L, Anyaneji, UJ, Tshiabuila, D, Viegas, S, Lessells, R, Engelbrecht, S, Gudo, E, Jani, I, Niemann, S, Wilkinson, E & de Oliveira, T 2023, 'Genomic epidemiology of SARS-CoV-2 during the first four waves in Mozambique', PLOS global public health, Jg. 3, Nr. 3, S. e0001593. https://doi.org/10.1371/journal.pgph.0001593
Koehler, N, Andres, S, Merker, M, Dreyer, V, John, A, Kuhns, M, Krieger, D, Choong, E, Verougstraete, N, Zur Wiesch, PA, Wicha, SG, König, C, Kalsdorf, B, Sanchez Carballo, PM, Schaub, D, Werngren, J, Schön, T, Peloquin, CA, Schönfeld, N, Verstraete, AG, Decosterd, LA, Aarnoutse, R, Niemann, S, Maurer, FP & Lange, C 2023, 'Pretomanid-Resistant Tuberculosis', Journal of infection, Jg. 86, Nr. 5, S. 520-524. https://doi.org/10.1016/j.jinf.2023.01.039
Noroc, E, Chesov, D, Merker, M, Gröschel, MI, Barilar, I, Dreyer, V, Ciobanu, N, Reimann, M, Crudu, V & Lange, C 2023, 'Limited Nosocomial Transmission of Drug-Resistant Tuberculosis, Moldova', EMERGING INFECTIOUS DISEASES, Jg. 29, Nr. 5, S. 1046-1050. https://doi.org/10.3201/eid2905.230035
Pérez-Llanos, FJ, Dreyer, V, Barilar, I, Utpatel, C, Kohl, TA, Murcia, MI, Homolka, S, Merker, M & Niemann, S 2023, 'Transmission Dynamics of a Mycobacterium tuberculosis Complex Outbreak in an Indigenous Population in the Colombian Amazon Region', Microbiology spectrum, S. e0501322. https://doi.org/10.1128/spectrum.05013-22
Rollo, RF, Mori, G, Hill, TA, Hillemann, D, Niemann, S, Homolka, S, Fairlie, DP & Blumenthal, A 2023, 'Wollamide Cyclic Hexapeptides Synergize with Established and New Tuberculosis Antibiotics in Targeting Mycobacterium tuberculosis', Microbiology spectrum, Jg. 11, Nr. 4, S. e0046523. https://doi.org/10.1128/spectrum.00465-23
Schlanderer, J, Hoffmann, H, Lüddecke, J, Golubov, A, Grasse, W, Kindler, EV, Kohl, TA, Merker, M, Metzger, C, Mohr, V, Niemann, S, Pilloni, C, Plesnik, S, Raya, B, Shresta, B, Utpatel, C, Zengerle, R, Beutler, M & Paust, N 2023, 'Two-stage tuberculosis diagnostics: combining centrifugal microfluidics to detect TB infection and Inh and Rif resistance at the point of care with subsequent antibiotic resistance profiling by targeted NGS', Lab on a chip, Jg. 24, Nr. 1, S. 74-84. https://doi.org/10.1039/d3lc00783a
Smith, JP, Modongo, C, Oeltmann, JE, Dima, M, Matsiri, O, Fane, O, Molefi, T, Shin, SS, Barilar, I, Niemann, S, Zetola, NM & Moonan, PK 2023, 'High-Resolution Characterization of Nosocomial Mycobacterium tuberculosis Transmission Events in Botswana', American journal of epidemiology, Jg. 192, Nr. 3, S. 503-506. https://doi.org/10.1093/aje/kwac214
Tagliani, E, Kohl, TA, Ghodousi, A, Groenheit, R, Holicka, Y, Niemann, S, Maurer, FP, Cirillo, DM & Cambau, E 2023, 'Appeal from the European reference laboratory network for tuberculosis for improving the diagnosis of infections caused by non-tuberculous mycobacteria', CLINICAL MICROBIOLOGY AND INFECTION . https://doi.org/10.1016/j.cmi.2023.06.005
TBNET and RESIST-TB networks, Domínguez, J, Boeree, MJ, Cambau, E, Chesov, D, Conradie, F, Cox, V, Dheda, K, Dudnyk, A, Farhat, MR, Gagneux, S, Grobusch, MP, Gröschel, MI, Guglielmetti, L, Kontsevaya, I, Lange, B, van Leth, F, Lienhardt, C, Mandalakas, AM, Maurer, FP, Merker, M, Miotto, P, Molina-Moya, B, Morel, F, Niemann, S, Veziris, N, Whitelaw, A, Horsburgh, CR & Lange, C 2023, 'Clinical implications of molecular drug resistance testing for Mycobacterium tuberculosis: a 2023 TBnet/RESIST-TB consensus statement', Lancet Infectious Diseases, Jg. 23, Nr. 4, S. e122-e137. https://doi.org/10.1016/S1473-3099(22)00875-1
Trauth, J, Matt, U, Kohl, TA, Niemann, S & Herold, S 2023, 'Blind spot in endocarditis guidelines: Mycobacterium chimaera prosthetic valve endocarditis after cardiac surgery-a case series', European heart journal. Case reports, Jg. 7, Nr. 8, S. ytad400. https://doi.org/10.1093/ehjcr/ytad400
Wetzstein, N, Kohl, TA, Diricks, M, Mas-Peiro, S, Holubec, T, Kessel, J, Graf, C, Koch, B, Herrmann, E, Vehreschild, MJGT, Hogardt, M, Niemann, S, Stephan, C & Wichelhaus, TA 2023, 'Clinical characteristics and outcome of Mycobacterium chimaera infections after cardiac surgery: systematic review and meta-analysis of 180 heater-cooler unit associated cases', CLINICAL MICROBIOLOGY AND INFECTION , Jg. 29, Nr. 8, S. 1008-1014. https://doi.org/10.1016/j.cmi.2023.03.005
Zwyer, M, Rutaihwa, LK, Windels, E, Hella, J, Menardo, F, Sasamalo, M, Sommer, G, Schmülling, L, Borrell, S, Reinhard, M, Dötsch, A, Hiza, H, Stritt, C, Sikalengo, G, Fenner, L, De Jong, BC, Kato-Maeda, M, Jugheli, L, Ernst, JD, Niemann, S, Jeljeli, L, Ballif, M, Egger, M, Rakotosamimanana, N, Yeboah-Manu, D, Asare, P, Malla, B, Dou, HY, Zetola, N, Wilkinson, RJ, Cox, H, Carter, EJ, Gnokoro, J, Yotebieng, M, Gotuzzo, E, Abimiku, A, Avihingsanon, A, Xu, ZM, Fellay, J, Portevin, D, Reither, K, Stadler, T, Gagneux, S & Brites, D 2023, 'Back-to-Africa introductions of Mycobacterium tuberculosis as the main cause of tuberculosis in Dar es Salaam, Tanzania', PLOS PATHOGENS, Jg. 19, Nr. 4, S. e1010893. https://doi.org/10.1371/journal.ppat.1010893
2022
Abdul, JBPAA, Adegbite, BR, Ndanga, MED, Edoa, JR, Mevyann, RC, Mfoumbi, GRAI, de Dieu, TJ, Mahoumbou, J, Biyogho, CM, Jeyaraj, S, Niemann, S, Lell, B, Kremsner, PG, Alabi, AS, Adegnika, AA & Grobusch, MP 2022, 'Resistance patterns among drug-resistant tuberculosis patients and trends-over-time analysis of national surveillance data in Gabon, Central Africa', Infection, S. 1-8. https://doi.org/10.1007/s15010-022-01941-5
Akwani, WC, van Vliet, AHM, Joel, JO, Andres, S, Diricks, M, Maurer, FP, Chambers, MA & Hingley-Wilson, SM 2022, 'The Use of Comparative Genomic Analysis for the Development of Subspecies-Specific PCR Assays for Mycobacterium abscessus', Frontiers in Cellular and Infection Microbiology, Jg. 12, S. 816615. https://doi.org/10.3389/fcimb.2022.816615
Antimycobacterial Susceptibility Testing Group 2022, 'Updating the approaches to define susceptibility and resistance to anti-tuberculosis agents: implications for diagnosis and treatment', The European respiratory journal, Jg. 59, Nr. 4, 2200166. https://doi.org/10.1183/13993003.00166-2022
Bateson, A, Ortiz Canseco, J, McHugh, TD, Witney, AA, Feuerriegel, S, Merker, M, Kohl, TA, Utpatel, C, Niemann, S, Andres, S, Kranzer, K, Maurer, FP, Ghodousi, A, Borroni, E, Cirillo, DM, Wijkander, M, Toro, JC, Groenheit, R, Werngren, J, Machado, D, Viveiros, M, Warren, RM, Sirgel, F, Dippenaar, A, Köser, CU, Sun, E & Timm, J 2022, 'Ancient and recent differences in the intrinsic susceptibility of Mycobacterium tuberculosis complex to pretomanid', JOURNAL OF ANTIMICROBIAL CHEMOTHERAPY, Jg. 77, Nr. 6, S. 1685-1693. https://doi.org/10.1093/jac/dkac070
Brandenburg, J, Heyckendorf, J, Marwitz, F, Zehethofer, N, Linnemann, L, Gisch, N, Karaköse, H, Reimann, M, Kranzer, K, Kalsdorf, B, Sanchez-Carballo, P, Weinkauf, M, Scholz, V, Malm, S, Homolka, S, Gaede, KI, Herzmann, C, Schaible, UE, Hölscher, C, Reiling, N & Schwudke, D 2022, 'Tuberculostearic Acid-Containing Phosphatidylinositols as Markers of Bacterial Burden in Tuberculosis', ACS infectious diseases, Jg. 8, Nr. 7, S. 1303-1315. https://doi.org/10.1021/acsinfecdis.2c00075
Chesov, E, Chesov, D, Maurer, FP, Andres, S, Utpatel, C, Barilar, I, Donica, A, Reimann, M, Niemann, S, Lange, C, Crudu, V, Heyckendorf, J & Merker, M 2022, 'Emergence of bedaquiline resistance in a high tuberculosis burden country', The European respiratory journal, Jg. 59, Nr. 3, 2100621. https://doi.org/10.1183/13993003.00621-2021
Claassens, M, Dreyer, V, Nepolo, E, Mokomele, Q, van Rooyen, G, Ruswa, N, Günther, G & Niemann, S 2022, 'Whole-Genome Sequencing for Resistance Prediction and Transmission Analysis of Mycobacterium tuberculosis Complex Strains from Namibia', Microbiology spectrum, Jg. 10, Nr. 5, S. e0158622. https://doi.org/10.1128/spectrum.01586-22
CRyPTIC Consortium 2022, 'A data compendium associating the genomes of 12,289 Mycobacterium tuberculosis isolates with quantitative resistance phenotypes to 13 antibiotics', PLOS BIOLOGY , Jg. 20, Nr. 8, S. e3001721. https://doi.org/10.1371/journal.pbio.3001721
CRyPTIC Consortium 2022, 'High fluoroquinolone resistance proportions among multidrug-resistant tuberculosis driven by dominant L2 Mycobacterium tuberculosis clones in the Mumbai Metropolitan Region', Genome medicine, Jg. 14, Nr. 1, S. 95. https://doi.org/10.1186/s13073-022-01076-0
CRyPTIC Consortium, Barilar, I, Dreyer, V, Kohl, T, Merker, M, Niemann, S & Utpatel, C 2022, 'The 2021 WHO catalogue of Mycobacterium tuberculosis complex mutations associated with drug resistance: A genotypic analysis', The Lancet. Microbe, Jg. 3, Nr. 4, S. e265-e273. https://doi.org/10.1016/S2666-5247(21)00301-3
CRyPTIC Consortium, Niemann, S, Barilar, I, Dreyer, V, Merker, M, Kohl, T & Utpatel, C 2022, 'Epidemiological cutoff values for a 96-well broth microdilution plate for high-throughput research antibiotic susceptibility testing of M. tuberculosis', The European respiratory journal, Jg. 60, Nr. 4, 2200239. https://doi.org/10.1183/13993003.00239-2022
Diricks, M, Kohl, TA, Käding, N, Leshchinskiy, V, Hauswaldt, S, Jiménez Vázquez, O, Utpatel, C, Niemann, S, Rupp, J & Merker, M 2022, 'Whole genome sequencing-based classification of human-related Haemophilus species and detection of antimicrobial resistance genes', Genome medicine, Jg. 14, Nr. 1, S. 13. https://doi.org/10.1186/s13073-022-01017-x
Diricks, M, Merker, M, Wetzstein, N, Kohl, TA, Niemann, S & Maurer, FP 2022, 'Delineating Mycobacterium abscessus population structure and transmission employing high-resolution core genome multilocus sequence typing', Nature communications, Jg. 13, Nr. 1, S. 4936. https://doi.org/10.1038/s41467-022-32122-5
European Laboratory Initiative on TB, HIV and Viral Hepatitis(3) & Niemann, S 2022, 'Diagnostic Capacities for Multidrug-Resistant Tuberculosis in the World Health Organization European Region: Action is Needed by all Member States', Journal of Molecular Diagnostics, Jg. 24, Nr. 11, S. 1189-1194. https://doi.org/10.1016/j.jmoldx.2022.07.005
Finci, I, Albertini, A, Merker, M, Andres, S, Bablishvili, N, Barilar, I, Cáceres, T, Crudu, V, Gotuzzo, E, Hapeela, N, Hoffmann, H, Hoogland, C, Kohl, TA, Kranzer, K, Mantsoki, A, Maurer, FP, Nicol, MP, Noroc, E, Plesnik, S, Rodwell, T, Ruhwald, M, Savidge, T, Salfinger, M, Streicher, E, Tukvadze, N, Warren, R, Zemanay, W, Zurek, A, Niemann, S & Denkinger, CM 2022, 'Investigating resistance in clinical Mycobacterium tuberculosis complex isolates with genomic and phenotypic antimicrobial susceptibility testing: a multicentre observational study', The Lancet. Microbe, Jg. 3, Nr. 9, S. e672-e682. https://doi.org/10.1016/S2666-5247(22)00116-1
Gisch, N, Utpatel, C, Gronbach, LM, Kohl, TA, Schombel, U, Malm, S, Dobos, KM, Hesser, DC, Diel, R, Götsch, U, Gerdes, S, Shuaib, YA, Ntinginya, NE, Khosa, C, Viegas, S, Kerubo, G, Ali, S, Al-Hajoj, SA, Ndung’u, PW, Rachow, A, Hoelscher, M, Maurer, FP, Schwudke, D, Niemann, S, Reiling, N & Homolka, S 2022, 'Sub-Lineage Specific Phenolic Glycolipid Patterns in the Mycobacterium tuberculosis Complex Lineage 1', Frontiers in Microbiology, Jg. 13, S. 832054. https://www.frontiersin.org/article/10.3389/fmicb.2022.832054
Goldstein, IH, Bayer, D, Barilar, I, Kizito, B, Matsiri, O, Modongo, C, Zetola, NM, Niemann, S, Minin, VM & Shin, SS 2022, 'Using genetic data to identify transmission risk factors: Statistical assessment and application to tuberculosis transmission', PLoS Computational Biology , Jg. 18, Nr. 12, S. e1010696. https://doi.org/10.1371/journal.pcbi.1010696
Große, C, Kohl, TA, Niemann, S, Herzberg, M & Nies, DH 2022, 'Loss of mobile genomic islands in metal resistant, hydrogen-oxidizing Cupriavidus metallidurans', APPLIED AND ENVIRONMENTAL MICROBIOLOGY , Jg. 88, Nr. 4, S. e0204821. https://doi.org/10.1128/AEM.02048-21
Hansen, J, Kolbe, K, König, IR, Scherließ, R, Hellfritzsch, M, Malm, S, Müller-Loennies, S, Zallet, J, Hillemann, D, Wiesmüller, K-H, Herzmann, C, Brandenburg, J & Reiling, N 2022, 'Lipobiotin-capture magnetic bead assay for isolation, enrichment and detection of Mycobacterium tuberculosis from saliva', PLOS ONE, Jg. 17, Nr. 7, S. e0265554. https://doi.org/10.1371/journal.pone.0265554
Kerubo, G, Ndungu, P, Shuaib, YA, Amukoye, E, Revathi, G, Homolka, S, Kariuki, S, Merker, M & Niemann, S 2022, 'Molecular Epidemiology of Mycobacterium tuberculosis Complex Strains in Urban and Slum Settings of Nairobi, Kenya', Genes, Jg. 13, Nr. 3, 475. https://doi.org/10.3390/genes13030475
Klein, C, Borsche, M, Balck, A, Föh, B, Rahmöller, J, Peters, E, Knickmann, J, Lane, M, Vollstedt, E-J, Elsner, SA, Käding, N, Hauswaldt, S, Lange, T, Hundt, JE, Lehrian, S, Giese, J, Mischnik, A, Niemann, S, Maurer, F, Homolka, S, Paulowski, L, Kramer, J, Twesten, C, Sina, C, Gillessen-Kaesbach, G, Busch, H, Ehlers, M, Taube, S, Rupp, J & Katalinic, A 2022, 'One-year surveillance of SARS-CoV-2 transmission of the ELISA cohort: A model for population-based monitoring of infection risk', Science advances, Jg. 8, Nr. 15, S. eabm5016. https://doi.org/10.1126/sciadv.abm5016
Mbelele, PM, Utpatel, C, Sauli, E, Mpolya, EA, Mutayoba, BK, Barilar, I, Dreyer, V, Merker, M, Sariko, ML, Swema, BM, Mmbaga, BT, Gratz, J, Addo, KK, Pletschette, M, Niemann, S, Houpt, ER, Mpagama, SG & Heysell, SK 2022, 'Whole genome sequencing-based drug resistance predictions of multidrug-resistant Mycobacterium tuberculosis isolates from Tanzania', JAC-antimicrobial resistance, Jg. 4, Nr. 2, S. dlac042. https://doi.org/10.1093/jacamr/dlac042
Merker, M, Rasigade, J-P, Barbier, M, Cox, H, Feuerriegel, S, Kohl, TA, Shitikov, E, Klaos, K, Gaudin, C, Antoine, R, Diel, R, Borrell, S, Gagneux, S, Nikolayevskyy, V, Andres, S, Crudu, V, Supply, P, Niemann, S & Wirth, T 2022, 'Transcontinental spread and evolution of Mycobacterium tuberculosis W148 European/Russian clade toward extensively drug resistant tuberculosis', Nature communications, Jg. 13, Nr. 1, S. 5105. https://doi.org/10.1038/s41467-022-32455-1
Mesfin, EA, Merker, M, Beyene, D, Tesfaye, A, Shuaib, YA, Addise, D, Tessema, B & Niemann, S 2022, 'Prediction of drug resistance by Sanger sequencing of Mycobacterium tuberculosis complex strains isolated from multidrug resistant tuberculosis suspect patients in Ethiopia', PLOS ONE, Jg. 17, Nr. 8, S. e0271508. https://doi.org/10.1371/journal.pone.0271508
Rachow, A, Saathoff, E, Mindru, R, Popescu, O, Lugoji, D, Mahler, B, Merker, M, Niemann, S, Olaru, ID, Kastner, S, Hoelscher, M, Lange, C & Ibraim, E 2022, 'Diagnostic performance of the AID line probe assay in the detection of Mycobacterium tuberculosis and drug resistance in Romanian patients with presumed TB', PLOS ONE, Jg. 17, Nr. 8, S. e0271297. https://doi.org/10.1371/journal.pone.0271297
Shuaib, YA, Utpatel, C, Kohl, TA, Barilar, I, Diricks, M, Ashraf, N, Wieler, LH, Kerubo, G, Mesfin, EA, Diallo, AB, Al-Hajoj, S, Ndung'u, P, Fitzgibbon, MM, Vaziri, F, Sintchenko, V, Martinez, E, Viegas, SO, Zhou, Y, Azmy, A, Al-Amry, K, Godreuil, S, Varma-Basil, M, Narang, A, Ali, S, Beckert, P, Dreyer, V, Kabwe, M, Bates, M, Hoelscher, M, Rachow, A, Gori, A, Tekwu, EM, Sidze, LK, Jean-Paul, AA, Beng, VP, Ntoumi, F, Frank, M, Diallo, AG, Mboup, S, Tessema, B, Beyene, D, Khan, SN, Diel, R, Supply, P, Maurer, FP, Hoffmann, H, Niemann, S & Merker, M 2022, 'Origin and Global Expansion of Mycobacterium tuberculosis Complex Lineage 3', Genes, Jg. 13, Nr. 6, 990. https://doi.org/10.3390/genes13060990
Sibandze, DB, Kay, A, Dreyer, V, Sikhondze, W, Dlamini, Q, DiNardo, A, Mtetwa, G, Lukhele, B, Vambe, D, Lange, C, Glenn Dlamini, M, Ness, T, Mejia, R, Kalsdorf, B, Heyckendorf, J, Kuhns, M, Maurer, FP, Dlamini, S, Maphalala, G, Niemann, S & Mandalakas, A 2022, 'Rapid molecular diagnostics of tuberculosis resistance by targeted stool sequencing', Genome medicine, Jg. 14, Nr. 1, S. 52. https://doi.org/10.1186/s13073-022-01054-6
The CRyPTIC Consortium & Niemann, S 2022, 'Genome-wide association studies of global Mycobacterium tuberculosis resistance to 13 antimicrobials in 10,228 genomes identify new resistance mechanisms', PLOS BIOLOGY , Jg. 20, Nr. 8, S. e3001755. https://doi.org/10.1371/journal.pbio.3001755
Wetzstein, N, Diricks, M, Kohl, TA, Wichelhaus, TA, Andres, S, Paulowski, L, Schwarz, C, Lewin, A, Kehrmann, J, Kahl, BC, Dichtl, K, Hügel, C, Eickmeier, O, Smaczny, C, Schmidt, A, Zimmermann, S, Nährlich, L, Hafkemeyer, S, Niemann, S, Maurer, FP & Hogardt, M 2022, 'Molecular Epidemiology of Mycobacterium abscessus Isolates Recovered from German Cystic Fibrosis Patients', Microbiology spectrum, Jg. 10, Nr. 4, S. e0171422. https://doi.org/10.1128/spectrum.01714-22
Zooniverse Volunteer Community & Niemann, S 2022, 'A crowd of BashTheBug volunteers reproducibly and accurately measure the minimum inhibitory concentrations of 13 antitubercular drugs from photographs of 96-well broth microdilution plates', eLife, Jg. 11, e75046. https://doi.org/10.7554/eLife.75046
2025
Geng, X, Barilar, I, Kesamang, M, Tumedi, KA, Matsheka, M, Modongo, C, Minin, V, Niemann, S & Shin, SS, SARS-CoV-2 Evolution in an HIV-Endemic Setting: A Genomic Epidemiology Study from Botswana, 2025, medRxiv. https://doi.org/10.1101/2025.11.21.25340589
2024
Baker, CR, Barilar, I, de Araujo, LS, Parker, DM, Fornace, K, Moonan, PK, Oeltmann, JE, Tobias, JL, Minin, VM, Modongo, C, Zetola, NM, Niemann, S & Shin, SS, Using genomic epidemiology and geographic activity spaces to investigate tuberculosis outbreaks in Botswana, 2024, bioRxiv. https://doi.org/10.1101/2024.12.04.24318520
Leitung der Forschungsgruppe

Wissenschaftliches Projektmanagement

Wissenschaftliche Mitarbeiter:innen






Campus Charité Mitte (CCM) and Campus Virchow-Klinikum (CVK)
Charité – Universitätsmedizin Berlin
Charitéplatz 1, 10117 Berlin










Theodor-Stern-Kai 7, 60590 Frankfurt/Main, Germany
Technische Mitarbeiter:innen